Whole Genome Sequencing (WGS) 데이터 사용하여 대장암 특이적인 구조변이 및 융합 유전자
정보를 효과적으로 발굴하고 정확하게 검출하기 위하여, 본 연구실에서 개발한 ETCHING (Efficient deTection of CHromosomal rearrangements and
fusIoN Genes)을 사용함. 기존의 개발된
다수의 구조변이 발굴 프로그램들은 WGS 데이터를 사용하여 구조변이 정보를 발굴할 때 매우 느린 속도와
많은 false-positive 정보를 생산하는 문제점을 보였음. 이러한
단점을 보완하기 위하여 ETCHING에서는 정상 세포와 다수의 전장 유전체 (hg19, hg38, 한국인, 일본인 그리고 중국인 등) 정보 그리고 dbSNP 정보를 사용해 구조변이와 관련된 sequencing read만 선별하여 사용하는 전략을 개발하였음. 그림 1.
ETCHING의 workflow(a,
b)와 ETCHING 전략의 효과 (c) TCGA BRCA WGS 데이터, HCC1395 데이터, 그리고 SNUH multiple myeloma WGS 데이터 등의
다양한 데이터를 사용하여 성능 평가를 진행한 결과 ETCHING은 다른 구조변이 발굴 프로그램들보다
대략 15배 더 빠른 실행 속도와 준수한 recall 그리고
뛰어난 precision을 보였음. 본 연구실에서 개발한
ETCHING을 사용한다면, 대장암 WGS 데이터를 사용하여 임상적으로 의미 있는 양질의 구조변이 정보를 효과적으로 검출할 수 있을 것으로 기대됨. 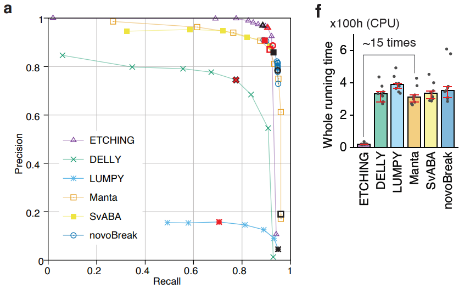
그림 2.
HCC1395 데이터를 사용한 성능 비교 평가에 대한 ROC curve와
TCGA BRCA WGS 데이터를 사용한 실행 속도 비교
평가 결과 |